Практикум 10
Поиск консервативных массивов в выравнивании
Для выполнения заданий данного практикума был выбран домен PHL domain с идентификатором PF20474. В выравнивании seed этого домена содержится 51 последовательность, в записях UniProt белков с данным доменом 478. PHL domain участвует в связывании липидов (например, фосфоинозитидов) или белковых взаимодействиях. Некоторые PHL-домены могут работать как каркасные элементы или регуляторные модули в сигнальных путях. PH-подобные домены (включая PHL) встречаются в киназах, адапторных белках и регуляторах GTPаз.
Выравнивание было скачено и помещено в Jalview. Идентичные последовательности были удалены (их не было). В Jalview была поставлена окраска Clustal, Above identity threshold 100% порог постепенно снижался, чтобы проявились мотивы. Нашлось достаточно много одиночных консервативных колонок и проявлялись некоторые мотивы. Наиболее консервативным на вид оказался мотив ADLLA на позициях 172-176.

Далее паттерн был переведен в формат Prosite: A-D-L-L-A
По этому мотиву был произведен поиск. В результате было найдено 1007 совпадений в 1000 последовательностях. Все совпадения получились идеальными, среди организмов есть как прокариоты, так и эукариоты. Можно сделать вывод, что результат не имеет смысла.
Поиск мотива, специфичного для одной клады филогенетического дерева
В Jalview было построено филогенетическое дерево для данного набора последовательностей.
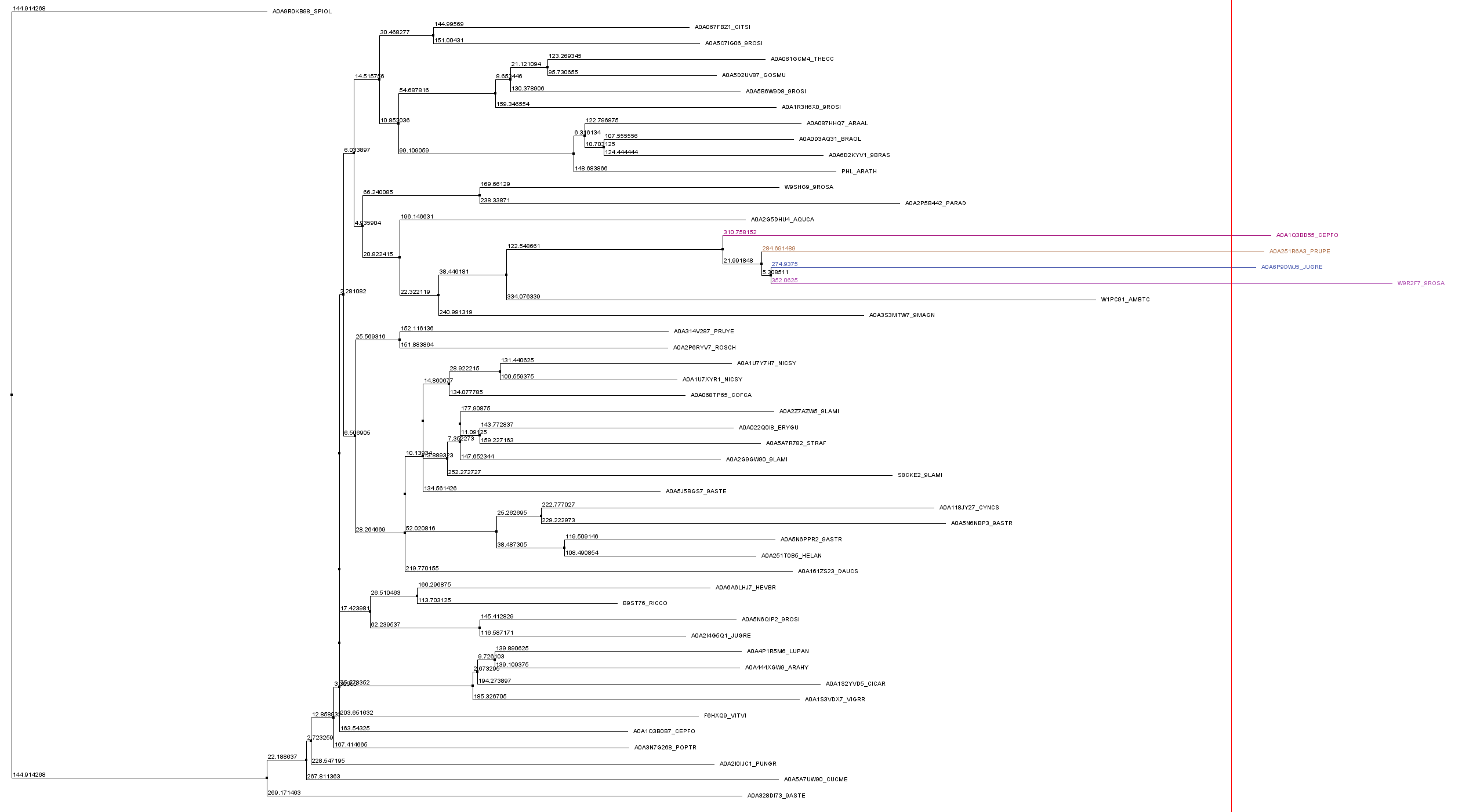
Затем была выбрана одна клада из 10 последовательностей. Для данной клады были повторены действия из первого задания, в результате было выделено несколько мотивов. Я выбрала мотив LSKS на позициях 51-55.

Данный мотив содержится в 35 последовательностях из 51, а клада состоит всего из 10 последовательностей. Получается, что это не специфичный для данной клады мотив. Мотив KTR (55-58) встречается в 28 последовательностях, мотив KVD (11-13) в 27, а мотив DGT (120-123) - в 39. Таким образом, мотив, специфичный именно для данной клады найти не удалось.
PSI-BLAST
Был случайно выбран идентификатор AC Q67XL4. Это нехарактеризованный CRM-домен-содержащий белок At3g25440, локализованный в хлоропласте Arabidopsis thaliana. CRM-домены связывают малые ядерные РНК и рибозимные РНК, регулируя их стабильность и активность участвуют в сплайсинге и процессинге РНК.
Таблица 1. Результаты поиска PSI-BLAST
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки | |
|---|---|---|---|---|---|---|
| 1 | 18 | Q9FFU1.1 | 0.004 | Q9SL79.2 | 1.20E-02 | |
| 2 | 23 | P54454.1 | 2.00E-04 | 0 | 0 | |
| 3 | 25 | Q58068.1 | 2.00E-06 | 0 | 0 | |
| 4 | 25 | Q58068.1 | 2.00E-06 | Q898C7.1 | 0.007 |
Из таблицы можно сделать вывод, что данный белок имеет близкие гомологи в SwissProt. Имеет место высокая консервативность. После первой итерации blast перестал находить белки с E-value ниже порога, после третьей перестал находить новые последовательности. Все белки, найденные в первой итерации принадлежат организму Arabidopsis thaliana, также находятся в хлоропласте и выполняют схожие функции. Последние найденные белки с E-value выше порога принадлежат бактериям и являются возможнымм РНК-связывающимм белками (функции схожие).
Поиск мотивов de novo с помощью MEME
Из базы данных Pfam были скачаны последовательности белков с выбранным ранее доменом в формате fasta, отличные от seed. Чтобы сократить список в Jalview были удалены высокосходные последовательности:
Далее был произведен поиск мотивов с помощью команды:
meme p.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4
В результате было найдено 4 мотива, все с хорошим E-value, один из них содержит последовательность ADLLA, найденную для seed в задании 1.

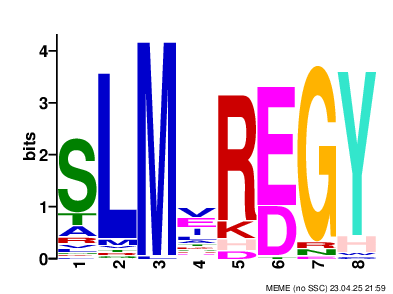
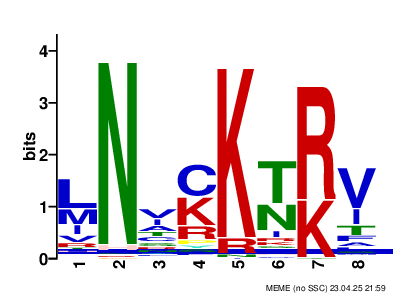

В результате запуска fimo командой
fimo results/meme.txt p.fastaбыло найдено 309 значимых появлений мотивов в 63 последовательностях. При поиске мотива, содержащего последовательность найденного мотива для seed командой
fimo --oc fimo_seed -motif ADLLAAQF -thresh 0.001 results/meme.txt p.fastaбыло найдено 75 значимых появлений. Это 24% от всех появлений мотивов. Отсюда можно предположить, что встречаемость всех мотивов примерно равна, и что мотив из seed не является наиболее часто встречающимся.
Оценка представленности сайта GATC в геноме археи Succinvibrio dextrinosolvens
Далее запускаем команду
cbcalc -s sites.txt -M -o result.tsv GCF_016747875.1_ASM1674787v1_genomic.fnaи по полученным данным строим диаграмму.
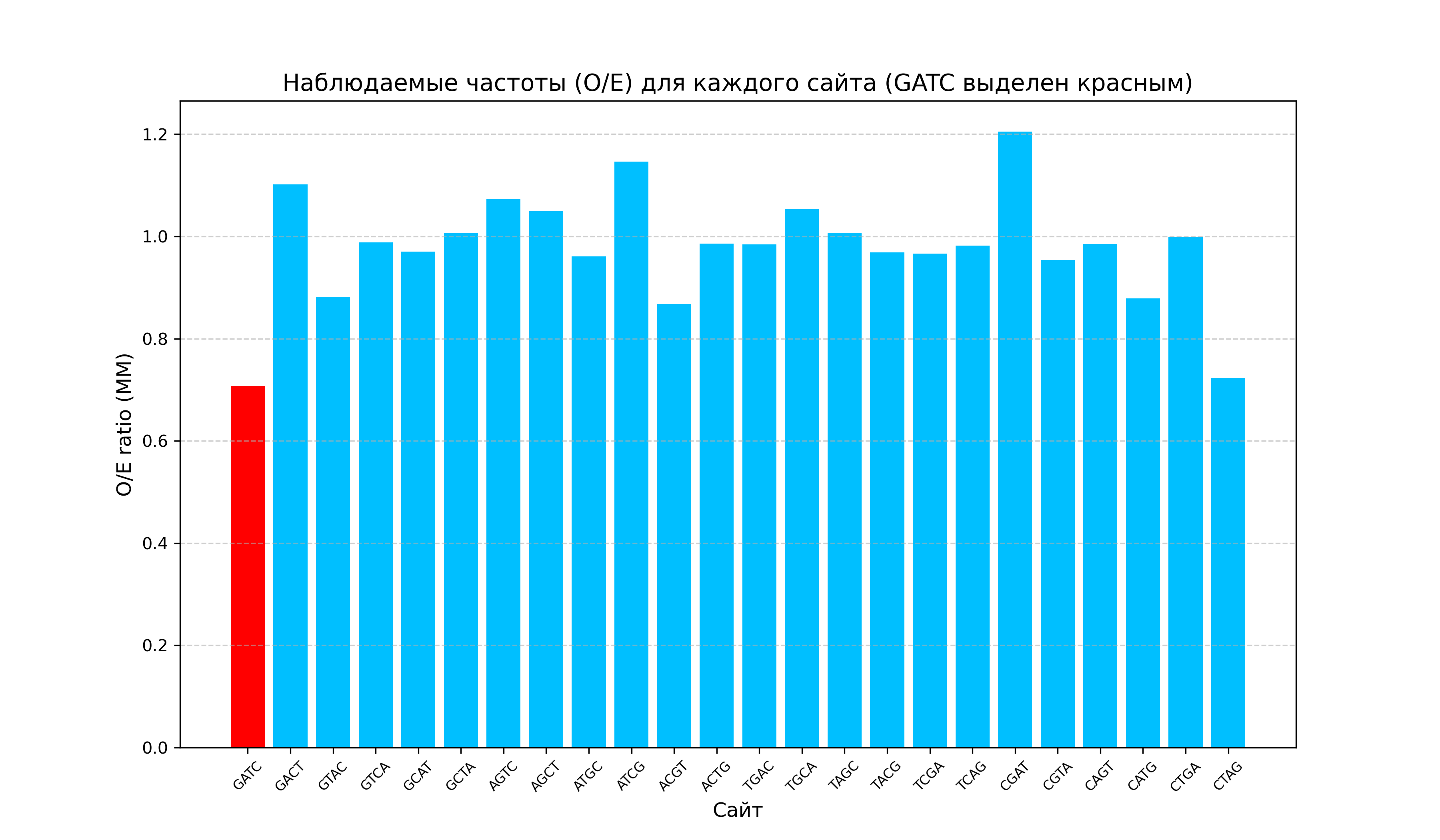
Из гистограммы явно видно, что сайт GATC представлен в наименьшем количестве у археи Succinvibrio dextrinosolvens. Интересно, что у 8 сайтов O/E примерно одинаковo, то есть ожидаемая частота совпадает с реальной. Можно предположить, что метилирование будет происходить по сайту CGAT или ATGC, так как они представлены больше ожидаемого количества.