Практикум 7
Задание 1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Название: Рецептор поглощения гидроксамата железа (Ferric hydroxamate uptake receptor FhuA)
Индификатор PDB:1qfg
Индификатор Uniprot:FHUA_ECOLI
Организм из которого был взят белок: Escherichia coli
Локализация:Внешняя мембрана грамотрицательных бактерий
Функции:
служит рецептором для фагов T5, φ80 и T1
является рецептором для колицина M (*белок с антибактериальной активностью, убивает другие бактерии того же или близких видов)
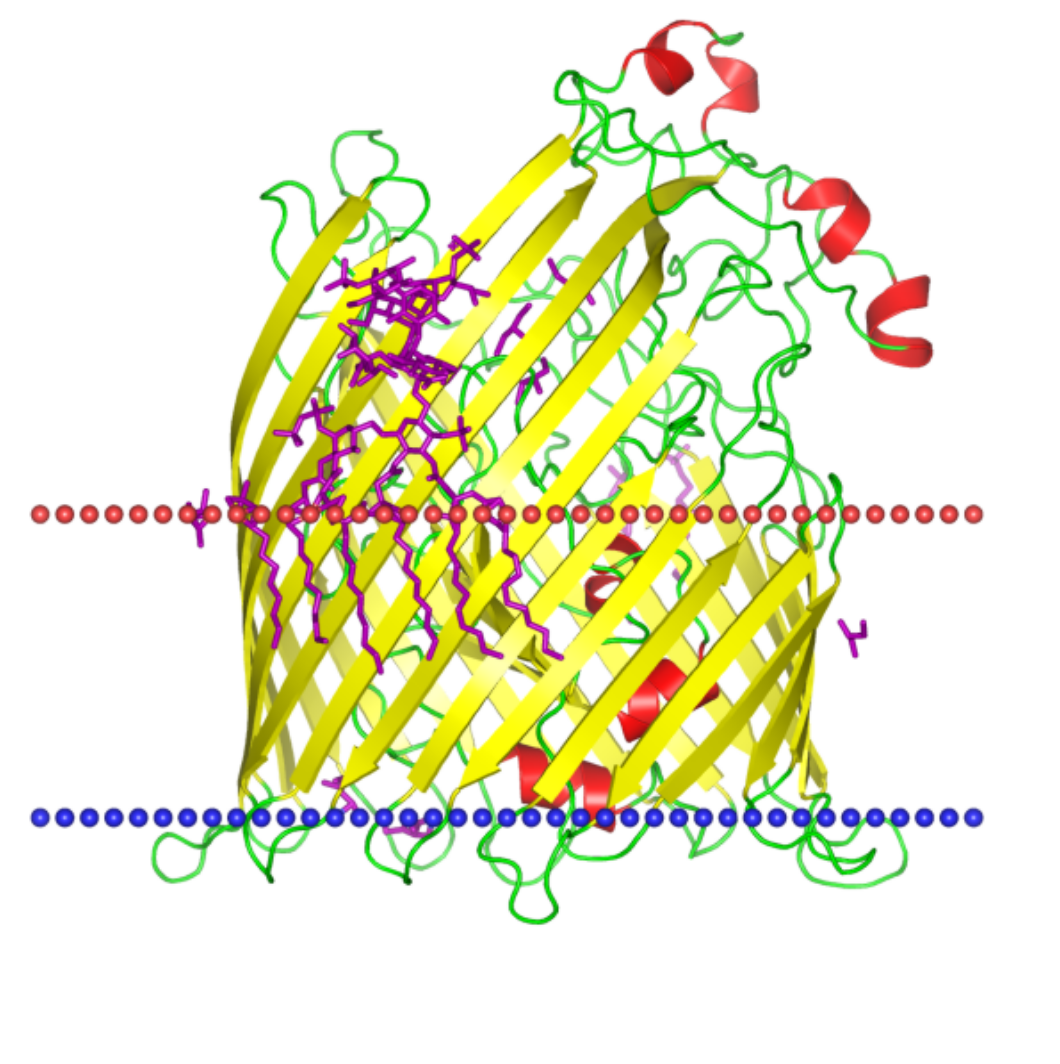
Координаты трансмембранных участков белка
A - Tilt: 1 - TM segments: 1(161-170), 2(173-182), 3(191-199), 4(212-221), 5(228-236), 6(280-288), 7(294-303), 8(355-365), 9(372-380), 10(443-453), 11(456-465), 12(487-497), 13(501-509), 14(531-540), 15(545-552), 16(580-589), 17(593-601),18(624-633), 19(641-649), 20(668-676), 21(685-692), 22(716-724)
DeepTMHMM
Для запуска белка в DeepTMHMM была взята его аминокислотная последовательность из PDB. Так как для предсказания в OPM используется зрелый белок, в котором нет сигнального пептида, то для предсказания DeepTMHMM будет логично взять ту же последовательность без сигнального пептида. Ее можно найти по ссылке: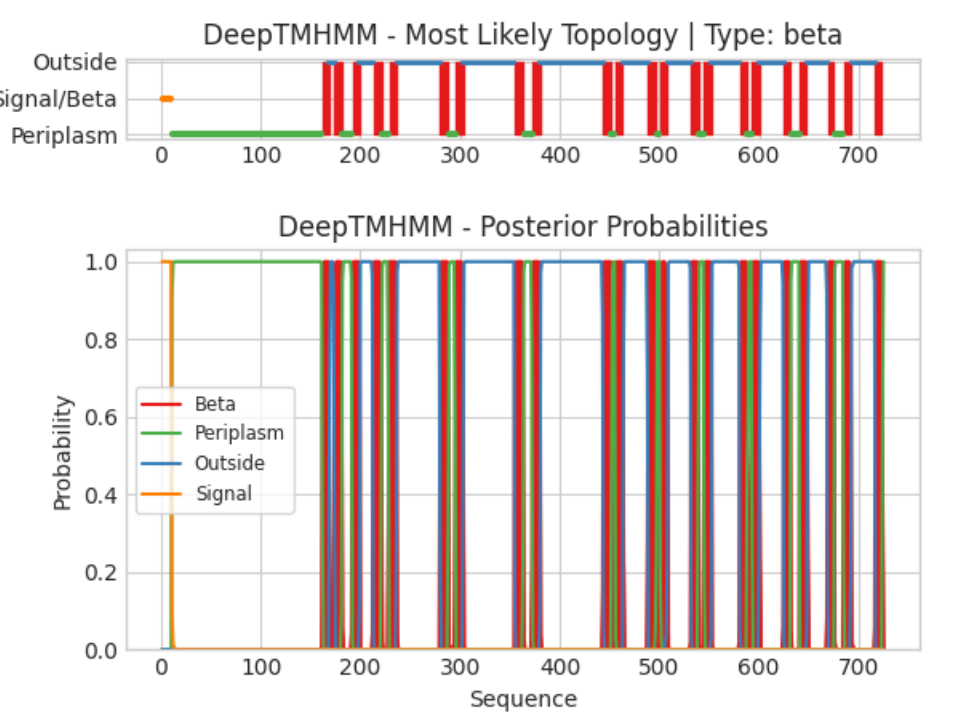
Предсказанные программой DeepTMHMM координаты трансмембранных бета-лисов:
Unnamed Beta sheet 162 168
Unnamed Beta sheet 174 181
Unnamed Beta sheet 192 199
Unnamed Beta sheet 213 221
Unnamed Beta sheet 229 236
Unnamed Beta sheet 280 288
Unnamed Beta sheet 296 303
Unnamed Beta sheet 355 363
Unnamed Beta sheet 373 380
Unnamed Beta sheet 444 451
Unnamed Beta sheet 456 463
Unnamed Beta sheet 489 497
Unnamed Beta sheet 501 508
Unnamed Beta sheet 532 539
Unnamed Beta sheet 545 552
Unnamed Beta sheet 581 587
Unnamed Beta sheet 593 600
Unnamed Beta sheet 625 631
Unnamed Beta sheet 641 648
Unnamed Beta sheet 669 675
Unnamed Beta sheet 685 692
Unnamed Beta sheet 717 723
Все предсказания DeepTMHMM, судя по нижнему графику, имеют высокую вероятность. Если сравнить предсказания OPM и DeepTMHMM, можно заметить, что обе программы предсказали по 22 β-листа (трансмембранных структур), 3 участка совпадают полностью (выделены зеленым), остальные участки сдвинуты на 1-2 а.к.о.
Вероятно, это происходит в силу различий в алгоритмах программ. DeepTMHMM анализирует только аминокислотную последовательность, а OPM учитывает трехмерную структуру белка, взятую из PDB. Поэтому неточное педсказание структуры может оказывать влияние на точность предсказания топологии.
Задание 2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке.
Название: UDP-N-ацетилглюкозамин-долихил-фосфат N-ацетилглюкозаминфосфотрансфераза (UDP-N-acetylglucosamine--dolichyl-phosphate N-acetylglucosaminephosphotransferase)
Индификатор PDB:6bw6
Индификатор Uniprot:GPT_HUMAN
Организм из которого был взят белок: Homo sapiens
Локализация:Мембрана эндоплазматического ретикулума
Функции:Катализирует первый этап биосинтеза долихол-связанных олигосахаридов - предшественников гликанов, используемых при N-гликозилировании белков.
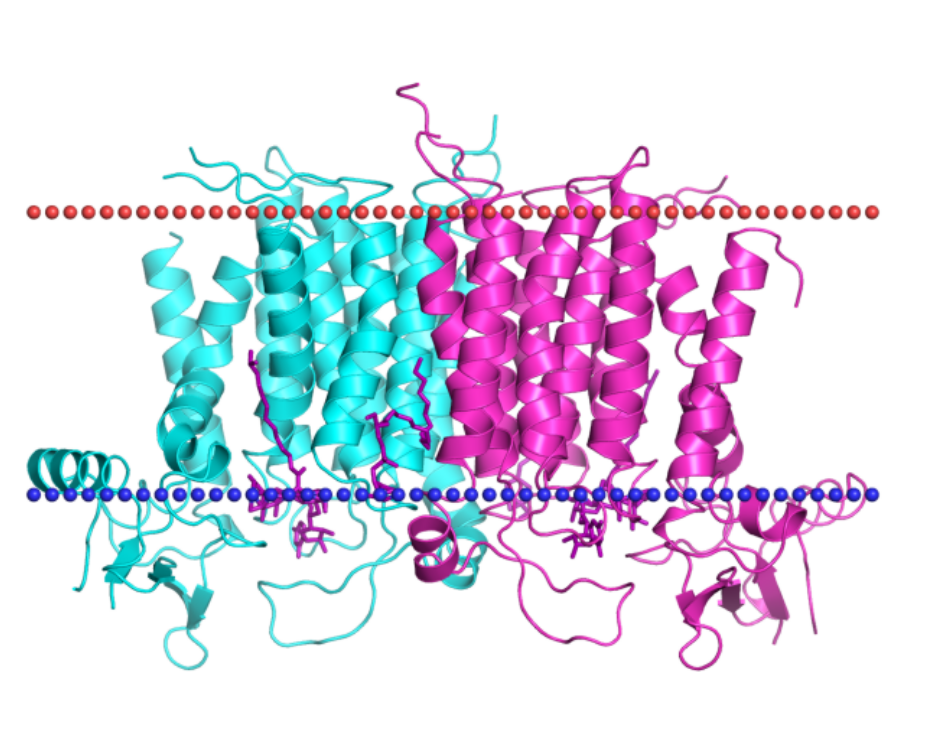
У данного белка 2 субъединицы, но цепь одна, координаты субъединиц A и B на сайте OPM совпадают:
A - Tilt: 6 - TM segments: 1( 10- 29), 2( 58- 78), 3( 95- 116), 4( 122- 140), 5( 167- 187), 6( 193- 212), 7( 220- 243), 8( 252- 269), 9( 272- 293),10( 380- 397)
Для запуска белка в DeepTMHMM была взята его аминокислотная последовательность из Uniprot. Ее можно найти по ссылке:
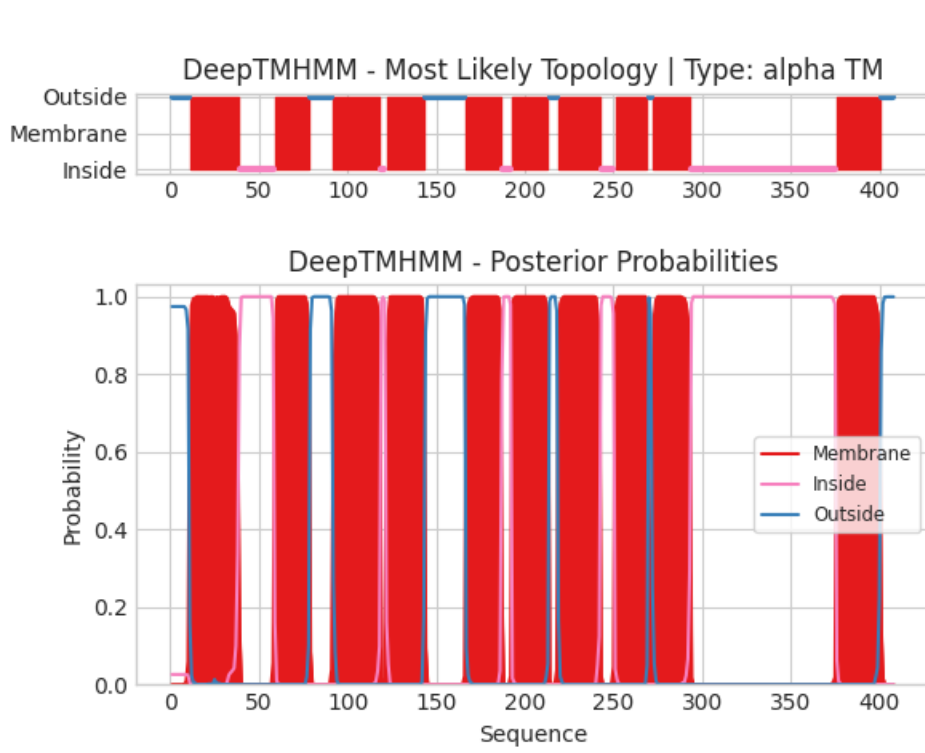
Обе программы предсказали по 10 трасмембранных структур. Координаты одной из них полностью совпадают, остальные незначительно отличаются (разница 1-2 а.к.о.) Такие небольшие различия в координатах структур могут объясняться постепенным переход от гидрофобной к гидрофильной среде, кроме того, белки могут иметь некоторую подвижность в мембране, что также может обосновать нечеткость границ.
Задание 3. TCDB.
Первый белок с AC P06971 имеет ТС-код TC #2.A.42.2.1, где
2 - класс: Транспортеры, управляемые электрохимическим потенциалом
А - Портеры (унипортеры, симпортеры, антипортеры)
транспортные системы включены в этот подкласс, если они используют процесс, опосредованный переносчиком, для катализа процессов унипорта (один вид переносится либо путем облегченной диффузии, либо в процессе, зависящем от мембранного потенциала, если растворитель заряжен), антипорта (два или более видов переносятся в противоположных направлениях в тесно связанном процессе, не связанном с прямой формой энергии, отличной от хемиосмотической энергии) и/или симпорта (два или более видов переносятся вместе в одном направлении в тесно связанном процессе, не связанном с прямой формой энергии, отличной от хемиосмотической энергии).
42 - Семейство гидрокси/ароматических аминокислотных пермеаз (HAAAP)
Семейство HAAAP включает три хорошо изученные ароматические аминокислоты: H+ симпортные пермеазы E. coli: высокоаффинную триптофан-специфическую пермеазу Mtr, низкоаффинную триптофан-пермеазу TnaB и тирозин-специфическую пермеазу TyrP, а также две хорошо изученные гидрокси-аминокислотные пермеазы - серин-пермеазу SdaC E. coli и треонин-пермеазу TdcC E. coli. Кроме того, в состав белка входит цистеиновая пермеаза CyuP (YhaO). Эти белки содержат 403-443 аминоацильных остатка и имеют одиннадцать предполагаемых или установленных ТМС. Все они участвуют в поглощении аминокислот. Гомологи присутствуют у большого числа грамотрицательных и грамположительных бактерий.
2 - Суперсемейство SdaC
Бактериальные транспортеры, связанные с переносом нуклеозидов или нуклеотидов.
1 - индивидуальнот для данного белка
Второй белок с АС Q9H3H5 в базе данных TCDB отсутствует.